Prokariotyczne systemy ekspresyjne
Dorota Porowińska 1 , Magdalena Wujak 2 , Katarzyna Roszek 2 , Michał Komoszyński 2Streszczenie
Do nadprodukcji rekombinowanych białek wykorzystywane są zarówno eukariotyczne, jak i prokariotyczne systemy ekspresyjne. Wybór odpowiedniego systemu zależy m.in. od tempa wzrostu komórek gospodarza i sposobu ich hodowli, poziomu ekspresji genu docelowego oraz obróbki potranslacyjnej syntetyzowanego białka. Niezależnie od rodzaju systemu ekspresyjnego, jego podstawowymi elementami są wektor i gospodarz ekspresyjny.
Najczęściej stosowanym układem do nadprodukcji białek, zarówno na skalę laboratoryjną jak i przemysłową, jest system prokariotyczny. System ten oparty jest przede wszystkim na bakteriach E. coli, chociaż coraz częściej wykorzystuje się bakterie z rodzaju Bacillus. System prokariotyczny pozwala na uzyskanie w krótkim czasie dużych ilości rekombinowanych białek. Prosta i tania hodowla komórek bakterii oraz dobrze poznane mechanizmy transkrypcji i translacji ułatwiają zastosowanie tych mikroorganizmów. Łatwość modyfikacji genetycznych i dostępność wielu mutantów bakteryjnych jest dodatkowym atutem systemu prokariotycznego. W pracy scharakteryzowano elementy strukturalne prokariotycznych wektorów ekspresyjnych. Omówiono także strategie przygotowania genu białka docelowego zwiększające wydajność jego produkcji, ułatwiające detekcję oraz oczyszczanie rekombinowanego białka i nadające mu aktywność. Scharakteryzowano szczepy bakteryjne najczęściej wykorzystywane jako komórki gospodarza w systemach ekspresyjnych oraz potencjalne umiejscowienie heterologicznego białka. Znajomość podstawowych elementów prokariotycznego systemu ekspresji pozwala na uzyskanie aktywnych biologicznie białek w krótkim czasie i w satysfakcjonujących ilościach.
Słowa kluczowe:nadprodukcja białek • wektor ekspresyjny • znacznik • E. coli • Bacillus
Summary
For overproduction of recombinant proteins both eukaryotic and prokaryotic expression systems are used. Choosing the right system depends, among other things, on the growth rate and culture of host cells, level of the target gene expression and posttranslational processing of the synthesized protein. Regardless of the type of expression system, its basic elements are the vector and the expression host.
The most widely used system for protein overproduction, both on a laboratory and industrial scale, is the prokaryotic system. This system is based primarily on the bacteria E. coli, although increasingly often Bacillus species are used. The prokaryotic system allows one to obtain large quantities of recombinant proteins in a short time. A simple and inexpensive bacterial cell culture and well-known mechanisms of transcription and translation facilitate the use of these microorganisms. The simplicity of genetic modifications and the availability of many bacterial mutants are additional advantages of the prokaryotic system. In this article we characterize the structural elements of prokaryotic expression vectors. Also strategies for preparation of the target protein gene that increase productivity, facilitate detection and purification of recombinant protein and provide its activity are discussed. Bacterial strains often used as host cells in expression systems as well as the potential location of heterologous proteins are characterized.
Knowledge of the basic elements of the prokaryotic expression system allows for production of biologically active proteins in a short time and in satisfactory quantities.
Key words:protein overproduction • expression vector • tag • E. coli • Bacillus
Wykaz skrótów:
CBP – peptyd wiążący kalmodulinę (calmodulin-binding peptide); Dsb -oksydoreduktazy tiolowo-disiarczkowe (thiol-oxidoreductases); fMET – formylometionina; FMN – mononukleotyd flawinowy; GFP – białko zielonej fluorescencji (green fluorescent protein); GST – transferaza-S-glutationu (glutathione S-transferase); IPTG – izopropylo-ß-D-galaktopiranozyd; LPS – lipopolisacharyd; MBP – białko wiążące maltozę (maltose-binding protein); MCS – miejsce wielokrotnego klonowania, polilinker (multiple cloning site); ori – miejsce inicjacji replikacji (origin of replication); RBS – sekwencja wiązania rybosomu (ribosomal binding site); SD – sekwencja Shine-Dalgarno; Strep – streptawidyna; tag – znacznik, etykieta; TrxA – tioredoksyna.
Wstęp
Dostępnych jest wiele różnych systemów pozwalających na nadprodukcję rekombinowanych białek. W tym celu wykorzystywane są zarówno komórki eukariotyczne jak i prokariotyczne. Wybór odpowiedniego systemu ekspresyjnego zależy od sposobu hodowli komórek, poziomu ekspresji, lokalizacji rekombinowanego białka oraz jego modyfikacji potranslacyjnych i aktywności biologicznej.
Komórki ssaków i owadów pozwalają na uzyskanie aktywnych katalitycznie cząsteczek mających wszystkie niezbędne modyfikacje potranslacyjne, jednak hodowla tych komórek jest czasochłonna i bardzo kosztowna, a ilość uzyskanego produktu jest przeważnie niewielka. W związku z tym zastosowanie tego rodzaju systemów ogranicza się głównie do badań laboratoryjnych. W przeciwieństwie do komórek eukariotycznych, hodowla komórek prokariotycznych jest tania i pozwala na uzyskanie w krótkim czasie dużych ilości rekombinowanych białek [16,17,21,26,27,28]. Ponadto komórki bakteryjne są często stosowane do produkcji heterologicznych białek ze względu na bardzo dobrze poznany mechanizm transkrypcyjny i translacyjny, szeroką dostępność promotorów i replikonów oraz możliwość zastosowania wielu różnych mutantów [16,17,19,28]. Wadą bakterii jest jednak ich niezdolność do przeprowadzania modyfikacji potranslacyjnych w syntetyzowanych białkach.
Niezależnie od rodzaju systemu ekspresyjnego oraz właściwości naprodukowanego białka, każdy system ekspresyjny składa się z dwóch elementów:
• wektora zawierającego gen i
• gospodarza.
Wektory ekspresyjne
Plazmidy to pozachromosomowe koliste cząsteczki DNA zbudowane przeważnie z dwóch nici. Występują naturalnie w cytoplazmie komórek bakteryjnych. Wykazują zdolność do samodzielnej replikacji niezależnie od chromosomu bakteryjnego dzięki obecności sekwencji ori [4].
Rozwój inżynierii genetycznej pozwolił na wykorzystanie tych autonomicznych cząsteczek do produkcji rekombinowanych białek użytecznych w przemyśle i medycynie. Dostępnych jest wiele różnych wektorów ekspresyjnych, których wybór możemy dostosować do własnych potrzeb. Niezależnie jednak od rodzaju rekombinowanego białka, które chcemy produkować, każdy wektor ekspresyjny powinien zawierać następujące elementy:
• replikon, • miejsce wielokrotnego klonowania (polilinker),
• promotor,
• miejsce wiązania rybosomu,
• terminator transkrypcji (ryc. 1) [19,26].
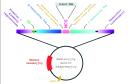
Ryc. 1. Schemat bakteryjnego wektora ekspresyjnego
Replikon
Replikon to fragment sekwencji plazmidu zawierający miejsce inicjacji replikacji (ori), umożliwiające samodzielne namnażanie plazmidu w komórce gospodarza. Wybór replikonu determinuje liczbę kopii wektora w komórce bakteryjnej. Do ekspresji rekombinowanych białek stosuje się zarówno plazmidy nisko-, średnio-, jak i wysokokopijne. Najczęściej wybierane są jednak replikony dające kilka do kilkunastu kopii plazmidu. Nie powoduje to zbyt dużego obciążenia metabolizmu komórki [9,16,28].
Większość wektorów ekspresyjnych ma replikon ColE1 lub p15A [3,16,20,24,28]. Ten pierwszy stosowany jest w wektorach pochodnych pBR322 (np. wektory pET, dające 15-20 kopii/komórkę) oraz wektorze pUC i jego pochodnych (500-700 kopii/komórkę). Replikon p15A pochodzi z plazmidu pACYC184 i warunkuje 10-12 kopii wektora w komórce [24]. W przypadku białek bardzo toksycznych, gdzie niezbędna jest ścisła kontrola ekspresji heterologicznych genów, stosowane są elementy replikonu episomu F (oriS, repF, parABC). Odpowiedzialne są one za rozdział plazmidów do komórek potomnych i utrzymanie pojedynczej kopii na każdą komórkę [16,22].
Promotor
Do ekspresji rekombinowanych białek stosuje się bardzo wiele różnorodnych promotorów [16,26,28]. Przy wyborze promotora bierze się pod uwagę takie jego cechy, jak rodzaj, siła, możliwość kontroli ekspresji, sposób i koszt indukcji oraz kompatybilność z wieloma szczepami bakteryjnymi [18].
W zależności od rodzaju rekombinowanego białka (jego toksyczności dla komórek bakteryjnych) stosuje się promotory indukowane lub konstytutywne. W przypadku tych pierwszych, szczególną uwagę zwraca się na możliwość ścisłej kontroli ekspresji genów znajdujących się pod kontrolą danego promotora. Nawet niewielka ekspresja takiego genu przy braku induktora może zmniejszyć stabilność plazmidu, sprzyjać generowaniu mutacji w genie rekombinowanego białka (jeśli białko jest toksyczne). Taka podstawowa ekspresja nazywana jest „przeciekaniem promotora” (promotor leakage) [3,16,28] lub „przeciekaniem plazmidu” (plasmid leakage) [7]. W celu uniknięcia takiej niekontrolowanej ekspresji genów docelowych w niektórych systemach ekspresyjnych stosuje się dodatkowe mechanizmy zabezpieczające w postaci np. genów represorowych uniemożliwiających ekspresję genu z danego promotora [28].
Promotory konstytutywne pozwalają na ciągłą syntezę rekombinowanego białka w komórkach bakterii. Zwykle stosuje się je w połączeniu z jednoczesnym wydzielaniem syntetyzowanego białka do przestrzeni periplazmatycznej lub podłoża hodowlanego. Nie ogranicza to wzrostu bakterii [28].
W wektorach ekspresyjnych używa się głównie silnych promotorów pozwalających na wydajną produkcję heterologicznych białek nawet do 10-30% całkowitego białka w komórce [3,16,26,28].
Promotor powinien być łatwo indukowany. Powszechnie stosuje się zarówno sposoby fizyczne (np. zmiana temperatury hodowli), jak i chemiczne (np. IPTG). W przypadku induktorów chemicznych ważne jest, aby był to związek niedrogi, ogólnie dostępny i niewykazujący negatywnego wpływu na wzrost bakterii [24,26,28].
Przykładowe promotory stosowane w plazmidach ekspresyjnych scharakteryzowano i opisano w tabeli 1.
Tabela 1. Promotory stosowane w plazmidach ekspresyjnych

Najczęściej stosowanym bakteryjnym systemem ekspresyjnym jest system wykorzystujący polimerazę RNA faga T7, którego mechanizm opiera się na bazie operonu laktozowego (wektory typu pET). W systemie tym w genomie komórki bakteryjnej znajduje się tzw. fragment λDE3 pochodzący z genomu faga λ, w obrębie którego znajduje się gen polimerazy RNA faga T7 znajdujący się po kontrolą promotora lacUV5 oraz operator lacO. Gen docelowy natomiast znajduje się pod kontrolą promotora faga T7. W warunkach nieindukcyjnych ekspresja genu spod promotora lacUV5 jest hamowana poprzez związanie w miejscu operatora białka represorowego LacI, którego gen (lacI) naturalnie występuje w genomie bakteryjnym. Uniemożliwia to transkrypcję genu fagowej polimerazy RNA i syntezę enzymu, a tym samym transkrypcję genu docelowego klonowanego do wektora. Ekspresja genu docelowego następuje w obecności induktora (IPTG), który wiąże się z białkiem LacI powodując uwolnienie promotora lacUV5. Umożliwia to ekspresję polimerazy RNA faga T7 i tym samym genu docelowego [16,26,28].
System oparty o polimerazę RNA faga T7 umożliwia wydajną syntezę wielu rekombinowanych białek. Jego dużą wadą jest jednak wysoki poziom ekspresji podstawowej, który może w znacznym stopniu zmniejszyć efektywność syntezy białka docelowego. Istnieje jednak wiele sposobów umożliwiających ograniczenie podstawowej ekspresji genów docelowych. W przypadku niektórych wektorów typu pET operator lacO znajduje się również w pobliżu promotora genu docelowego (promotor hybrydowy T7/lac), co stanowi dodatkowe zabezpieczenia przed „przeciekaniem plazmidu”. Innym sposobem ścisłej kontroli ekspresji jest zastosowanie szczepów bakteryjnych mających wektory pLysS i pLysE, które zawierają geny kodujące lizozym T7 – inhibitor polimerazy RNA faga T7. Podstawową ekspresję genów znajdujących się w wektorze pET ogranicza się również poprzez zastosowanie katabolicznej represji przez dodanie 0,5-1% glukozy do pożywki bakteryjnej [16,26,28].
Marker selekcyjny
Każdy wektor ekspresyjny powinien mieć „mechanizm” umożliwiający łatwą i jednoznaczną selekcję komórek zawierających plazmid od bakterii, które nie uległy transformacji. Najczęściej stosowanymi markerami są geny determinujące nabycie oporności na antybiotyki (np. ampicylinę, kanamycynę, tetracyklinę i chloramfenikol) [26].
Plazmidy dające oporność na ampicylinę zawierają gen bla, którego ekspresja powoduje syntezę w komórce β-laktamazy. Enzym ten wydzielany jest do periplazmy, gdzie katalizuje hydrolizę pierścienia laktamowego ampicyliny znajdującej się w środowisku hodowlanym [24]. Wadą stosowania ampicyliny jako markera selekcyjnego jest jej mała stabilność w środowisku wodnym. Wzrost pH pożywki w czasie wzrostu bakterii do dużych gęstości powoduje nieenzymatyczną degradację antybiotyku. Może to zmniejszać selektywność transformantów i powodować zanieczyszczenia hodowli komórkami niezawierającymi plazmidu. Rozwiązaniem tego problemu jest stosowanie trudniej degradowalnego analogu ampicyliny – karbenicyliny [24,26].
Kanamycyna jest inaktywowana w periplazmie komórek przez fosfotransferazę aminoglikozydu. Tak więc oporność bakterii na ten antybiotyk wymaga obecności genu ntpII w plazmidzie. Degradacja chloramfenikolu natomiast zachodzi z udziałem produktu genu cat – transferazy acetylochloramfenikolu. Oporność bakterii na tetracyklinę może być warunkowana przez wiele różnych genów [24].
Selekcja antybiotykami może zostać znacznie ograniczona (zmniejszona) w wyniku ich degradacji lub inaktywacji bądź wycieku z periplazmy enzymów hydrolizujących antybiotyki. Ponadto zanieczyszczenie preparatu rekombinowanego białka antybiotykami stosowanymi do selekcji uniemożliwia jego wykorzystanie, jeśli białko to ma mieć zastosowanie terapeutyczne [9].
Sekwencja Shine-Dalgarno (SD)
Synteza białka na matrycy genu klonowanego do wektora wymaga obecności sekwencji Shine-Dalgarno, która naturalnie znajduje się w obrębie sekwencji wiązania rybosomu (RBS). Jest to kilkunukleotydowa sekwencja, która oddziałuje z 3′ końcem 16S rRNA małej podjednostki rybosomu podczas inicjacji translacji. SD powinna znajdować się w odległości 7±2 nukleotydów od kodonu start translacji (AUG) [20,26]. Zmniejszenie tej odległości do 4 lub zwiększenie do 14 nukleotydów znacząco wpływa na wydajność inicjacji translacji [20,26]. Wydajność translacji można dodatkowo wzmocnić sekwencją epsilon faga T7, zlokalizowaną powyżej sekwencji SD. Z kolei czynnikiem obniżającym poziom biosyntezy heterologicznego białka jest tworzenie się struktur drugorzędowych w transkrypcie mRNA w obrębie rejonu SD [3,16].
Miejsce wielokrotnego klonowania (MCS, polilinker)
Kolejnym elementem wektora ekspresyjnego jest rejon do klonowania insertu, tzw. polilinker lub miejsce wielokrotnego klonowania (MCS). W jego obrębie znajdują się sekwencje nukleotydowe (tzw. miejsca restrykcyjne) specyficznie rozpoznawane przez endonukleazy restrykcyjne. Miejsca te występują w wektorze tylko raz (tj. w obrębie polilinkera), co pozwala na insercję genu w ściśle określone miejsce wektora i we właściwej orientacji. Ponadto, w przypadku produkcji białek fuzyjnych (heterologicznych białek zaopatrzonych w jeden lub kilka znaczników), wybór odpowiednich miejsc restrykcyjnych umożliwia klonowanie insertu z zachowaniem tej samej ramki odczytu, co sekwencja nukleotydowa kodująca znacznik [19,20].
Terminator transkrypcji
Poniżej sekwencji heterologicznego genu powinien się znajdować silny terminator transkrypcji. Jest to zwykle palindromowa sekwencja tworząca strukturę spinki do włosów, po której następuje ciąg nukleotydów adenylowych. Powoduje to zatrzymanie i oddysocjowanie polimerazy RNA. Dodatkowo tworząca się struktura na 3′ końcu powstałego mRNA stabilizuje tę cząsteczkę i wydłuża okres jej półtrwania. Wydajność terminacji transkrypcji zwiększa się w E. coli stosując kilkakrotne powtórzenie kodonu stop lub tzw. przedłużony kodon stop (UAAU) [9,20,26].
Znaczniki
Znaczniki (tzw. tagi lub etykiety) to krótkie peptydy, domeny lub całe białka dołączone do rekombinowanego produktu. Mają one na celu ułatwienie oczyszczania, detekcję i/lub zwiększenie rozpuszczalności rekombinowanego białka. Stosowany znacznik nie powinien w żaden sposób zaburzać struktury przestrzennej białka, ani wpływać na jego aktywność katalityczną. Powinien być łatwo usuwany od rekombinowanego białka na każdym etapie jego izolacji [2,26,29].
Znaczniki mogą być usytuowane na N – lub C-końcu łańcucha polipeptydowego. Dołączenie znacznika na N-końcu białka wpływa na efektywną inicjację procesu translacji, co zwiększa wydajność jego syntezy. Niezależnie od miejsca usytuowania znacznika może on wpływać stabilizująco na łańcuch polipeptydowy w komórkach bakteryjnych, chroniąc przed jego degradacją [26,29,30].
Wybór odpowiedniego znacznika zależy od rodzaju i właściwości syntetyzowanych cząsteczek. W celu zwiększenia wydajności produkcji i oczyszczania aktywnych katalitycznie białek możliwe jest stosowanie jednocześnie dwóch różnych znaczników.
Znaczniki ułatwiające oczyszczanie
Podstawową funkcją znaczników jest ułatwianie oczyszczania białek syntetyzowanych w systemach ekspresyjnych. Mają one umożliwić otrzymywanie homogennych preparatów dzięki zastosowaniu wydajnej i wysoce specyficznej chromatografii powinowactwa. Proces ten powinien zachodzić na niedrogich, trwałych i odpornych złożach, nadających się do wielokrotnego użytku w różnych warunkach środowiska (pH, siła jonowa, obecność detergentów, związków redukujących itp.). Jednocześnie elucja białka powinna zachodzić w łagodnych warunkach niepowodujących jego denaturacji. Najczęściej stosowanym znacznikiem ułatwiającym oczyszczanie jest His-Tag (zob. tabela 2) [2,16,26,29].
Tabela 2. Znaczniki rekombinowanych białek

Znaczniki ułatwiające detekcję
Znacznikami ułatwiającymi detekcję mogą być znaczniki nadające rekombinowanemu białku barwę lub fluorescencję (tzw. rainbow tags). Umożliwiają monitorowanie lokalizacji białka w komórce oraz podczas kolejnych etapów jego oczyszczania. Muszą być one jednak stosowane w połączeniu z innymi znacznikami używanymi do oczyszczania. Do najczęściej stosowanych znaczników detekcyjnych należy domena reduktazy cytochromu P450 odpowiedzialna za wiązanie FMN, nadająca syntetyzowanemu białku kolor niebiesko-zielony lub żółty oraz czerwony cytochrom b5. Dużą zaletą tych znaczników jest to, że ich barwa jest widoczna gołym okiem. Nie wymagają zewnętrznego źródła energii do ich detekcji, w przeciwieństwie do równie często stosowanego białka GFP (tabela 2) [2, 26, 29].
Znaczniki zwiększające rozpuszczalność
Produkcja rekombinowanych białek w komórkach prokariotycznych często prowadzi do tworzenia nierozpuszczalnych agregatów. W celu rozwiązania lub przynajmniej ograniczenia tego problemu do białka dołączane są znaczniki zwiększające rozpuszczalność. Są to na ogół małe i średnie białka charakteryzujące się dobrą rozpuszczalnością pochodzące z E. coli. Tego typu znaczniki są przeważnie usytuowane na N-końcu rekombinowanego białka. Wiele z nich jest również wykorzystywanych do jego oczyszczania [6,16,22,26,29].
Wady i zalety najczęściej stosowanych znaczników w rekombinowanych białkach zostały przedstawione w tabeli 2.
Usuwanie znaczników
Znaczniki, mimo że są pomocne w oczyszczaniu rekombinowanych białek, mogą niekiedy wpływać na ich aktywność biologiczną. Ponadto obecność etykiety może zaburzać analizę jego właściwości czy struktury przestrzennej. W związku z tym po zakończeniu procesu oczyszczania (lub w jego trakcie) niezbędne jest usunięcie znacznika. W tym celu stosowane są swoiste proteazy (endopeptydazy). Do najczęściej stosowanych należą: enterokinaza, TEV, trombina oraz czynnik Xa (tabela 3) [1,10,29].
Tabela 3. Proteazy stosowane do odcinania znaczników rekombinowanych białek

Odcinanie znacznika nie jest procesem prostym i wymaga optymalizacji dla każdego białka. Niektóre proteazy pozostawiają 1-2 aminokwasy na N-końcu łańcucha białkowego lub 4-6 aa na końcu C, w zależności od umiejscowienia znacznika. Nawet taka niewielka ilość dodatkowych aminokwasów w sekwencji białka może zaburzać jego aktywność katalityczną lub wpływać na badania struktury. Rozwiązaniem są często mniej swoiste proteazy (np. czynnik Xa, enterokinaza), których cięcie pozwala na uzyskanie natywnego N-końca białka. Dodatkowo w przypadku znaczników zwiększających rozpuszczalność, jego usunięcie niesie ze sobą ryzyko wytrącenia białka z roztworu [26,29].
Gen białka docelowego
Ekspresja rekombinowanych białek wymaga nie tylko wyboru odpowiedniego wektora i gospodarza, ale także odpowiedniego przygotowania genu białka docelowego. Ma to szczególne znaczenie przy produkcji białek eukariotycznych w prokariotycznych systemach ekspresyjnych.
Używalność kodonów
Zgodnie z definicją kodu genetycznego jeden aminokwas może być kodowany przez kilka kodonów. Częstość wykorzystywania takich synonimicznych kodonów przez komórki prokariotyczne jest inna niż w komórkach eukariotycznych. Prowadzi to do różnic w puli dostępnych tRNA w zależności od organizmu [8,9,12,20]. Z tego powodu ekspresja heterologicznych białek w komórkach bakterii może być utrudniona lub wręcz niemożliwa. Występowanie rzadkich kodonów może prowadzić do pojawienia się błędów podczas translacji, takich jak substytucja, przesunięcie ramki odczytu, przeskok kodonów lub przedwczesna terminacja translacji [3,9,20,24].
Problem różnej używalności kodonów można rozwiązać poprzez zmianę kodonów rzadko występujących w bakteriach. Proces ten można przeprowadzić stosując mutagenezę specyficzną miejscowo lub projektując cały gen de novo. Ten ostatni jest procesem skomplikowanym i bardzo czasochłonnym. Poza wymianą rzadko używanych kodonów należy również wyeliminować sekwencje powtórzone, sekwencje mogące tworzyć struktury drugorzędowe w mRNA oraz sekwencje rozpoznawane przez restryktazy. W związku z tym do projektowania genów często stosuje się specjalne programy komputerowe (np. GeneDesigner) [20,24,26].
Najczęściej stosowanym rozwiązaniem różnej częstości wykorzystywania kodonów są szczepy bakteryjne niosące geny kodujące tRNA rzadko występujące w bakteriach (np. BL21 CodonPlus). Wprowadzenie dodatkowej puli takich cząsteczek tRNA zwiększa wydajność syntezy heterologicznych białek, jednak znacznie obciąża to metabolizm komórek. Dodatkowo, niewłaściwa modyfikacja produkowanych cząsteczek tRNA może skutkować umieszczeniem w łańcuchu białkowym nieprawidłowych aminokwasów. Innym sposobem może być zmiana warunków hodowli bakterii (skład pożywki, obniżenie temperatury). Jednak to rozwiązanie rzadko bywa skuteczne [20,24,26].
Stabilność rekombinowanego białka
Przygotowując gen rekombinowanego białka do jego nadekspresji w komórkach prokariotycznych, należy zwrócić uwagę na rodzaj aminokwasu występującego bezpośrednio po formylometioninie (fMet). Od niego zależy wydajność usuwania fMet z łańcucha polipeptydowego przez aminopeptydazę metioninową. Proces ten zachodzi z małą wydajnością (16%) jeśli po fMet znajduje się reszta His, Gln, Glu, Phe, Met, Lys, Tyr, Trp lub Arg. Okres półtrwania białka po odcięciu metioniny zależy od N-końcowej reszty aminokwasowej polipeptydu. Jeśli jest to Arg, Lys, Phe, Leu, Trp lub Tyr czas ten wynosi tylko 2 minuty. W przypadku pozostałych aminokwasów – 10 minut. Ponadto Leu znajdująca się na początku łańcucha polipeptydowego zwiększa ryzyko jego proteolitycznej degradacji w komórkach bakterii [16,26].
Na stabilność syntetyzowanych białek w komórkach bakterii ma wpływ również sekwencja aminokwasowa ich karboksylowego końca. Należy zwrócić uwagę, aby sekwencja ta nie była podobna do peptydu ANDENYALAA. Powstaje on na końcach łańcuchów polipeptydowych utworzonych na bazie uszkodzonego mRNA, który nie ma kodonu stop. Takie cząsteczki kierowane są na drogę proteolitycznej degradacji [16].
Komórki gospodarza
Dostępnych jest wiele szczepów bakteryjnych przystosowanych do produkcji różnych typów białek. Wybór odpowiedniego gospodarza będzie wpływał na wydajność produkcji oraz jakość rekombinowanego białka. Odpowiedni gospodarz powinien być przede wszystkim genetycznie kompatybilny ze stosowanym wektorem, aby produkcja heterologicznego białka była możliwa i jak najbardziej efektywna. Ważna jest również stabilność plazmidów w cytoplazmie komórek bakteryjnych. Ponadto stosowany szczep bakterii powinien być pozbawiony jak największej liczby endogennych proteaz, które mogą degradować syntetyzowane polipeptydy.
Escherichia coli
Escherichia coli to Gram-ujemna bakteria należąca do rodziny Enterobacteriaceae. Bakteria ta jest najczęściej stosowanym gospodarzem do produkcji rekombinowanych białek [1,28]. Powszechnie wykorzystuje się pochodne szczepów BL21 oraz K12 [26,28]. Podstawową zaletą E. coli jest dobrze scharakteryzowany genom, poznany układ transkrypcyjny i translacyjny oraz łatwość manipulacji genetycznych. Pozwoliło to na stworzenie wielu mutantów tych bakterii, dzięki czemu możliwe jest dobranie odpowiedniego szczepu w zależności od właściwości i budowy heterologicznych białek. Ponadto bakterie te są zdolne do szybkiego wzrostu do dużych gęstości na niedrogich podłożach [19,21]. Wszystkie szczepy ekspresyjne E. coli są całkowicie pozbawione czynników patogennych. Nie są one zdolne do przeżycia w tkankach ludzi oraz zwierząt. Wadą E. coli jest niezdolność do przeprowadzenia modyfikacji potranslacyjnych wymaganych przez wiele białek eukariotycznych, brak wydajnego systemu sekrecji produkowanych polipeptydów do pożywki [11,22] oraz ograniczona zdolność do tworzenia mostków dwusiarczkowych [21,24]. Dodatkowym problemem jest obecność w preparatach rekombinowanych białek lipopolisacharydu (LPS) – endotoksyny o właściwościach pirogennych. W przypadku białek o zastosowaniu terapeutycznym, konieczne jest całkowite usunięcie LPS, co wiąże się z przeprowadzeniem dodatkowych etapów oczyszczania [28].
Bacillus sp.
Poza E. coli najczęściej wybieranymi drobnoustrojami do produkcji rekombinowanch białek, są Gram-dodatnie bakterie z rodzaju Bacillus. Dostępnych jest wiele szczepów pozwalających na wydajną produkcję heterologicznych białek. Do najczęściej stosowanych należą: B. megaterium, B. subtilis, B. licheniformis oraz B. brevis [28]. Wykorzystywane w biotechnologii szczepy Bacillus nie są patogenne i nie wydzielają toksycznych metabolitów wtórnych, stąd są odpowiednimi producentami enzymów używanych w przetwórstwie żywności. Ich dużą zaletą jest brak lipopolisacharydu w zewnętrznej błonie oraz wydajny aparat sekrecyjny heterologicznych białek do podłoża, co niezwykle ułatwia ich izolację i oczyszczanie [14,31,32]. Ponadto, częstość wykorzystywania synonimicznych kodonów przez komórki Bacillus jest podobna jak w komórkach eukariotycznych, co ułatwia wydajną produkcję heterologicznych białek i wyraźnie wyraźnie eliminuje ryzyko wystąpienia błędów podczas ich syntezy [31]. Wadą tych bakterii jako systemów do ekspresji jest wymóg stosowania bogatszych podłoży hodowlanych, niższy poziom produkcji heterologicznego białka w porównaniu do komórek E. coli, niestabilność plazmidów, duża aktywność endogennych proteaz oraz zdolność do tworzenia spor w odpowiedzi na stres żywieniowy. Większość powyższych ograniczeń może zostać wyeliminowanych poprzez generowanie genetycznie zmodyfikowanych szczepów (mutantów) charakteryzujących się ograniczonym potencjałem do sporulacji i brakiem większości proteaz, a także przez stosowanie silnych promotorów oraz dodatkowych plazmidów niosących dodatkowe kopie genów kodujących białka opiekuńcze (chaperony) [31,32]. Chaperony (GroES, GroEL, DnaK, DnaJ, GrpE i PrsA) stanowią element aparatu sekrecyjnego i odpowiadają za poprawne fałdowanie białek, hamowanie ich agregacji, a także ich translokację przez błonę komórkową [31].
Proces sekrecji heterologicznych białek w bakteriach z rodzaju Bacillus obejmuje trzy fazy (ryc. 2). W pierwszej fazie następuje synteza heterologicznego białka w fuzji z sekwencją sygnałową oraz jego interakcja z chaperonami. Następnie związanie białka z translokazą sekrecyjną umożliwia jego transport w poprzek błony cytoplazmatycznej (faza druga). W trzeciej fazie następuje usunięcie sekwencji sygnałowej oraz ponowne fałdowanie białka w obecności chaperonów. Ostatnim etapem jest przeniknięcie poprawnie sfałdowanego białka przez ścianę komórkową i uwolnienie go do podłoża hodowlanego [14,31].

Ryc. 2. Możliwe losy rekombinowanego białka w komórkach bakterii z rodzaju Bacillus [zmodyfikowano wg 14]
Nadprodukowane w bakteriach białka mogą być akumulowane w cytoplazmie, periplazmie lub być wydzielane na zewnątrz komórki. Docelowa lokalizacja syntetyzowanego białka powinna zostać uwzględniona już na etapie wyboru wektora ekspresyjnego.
Cytoplazma
Cytoplazma komórek bakteryjnych jest najczęściej wybieraną lokalizacją do akumulacji rekombinowanych białek. Jest to związane głównie z bardzo dużą wydajnością produkcyjną w tym przedziale komórkowym. Białka akumulowane w cytoplazmie są jednak narażone na działanie endogennych proteaz. Przy wyborze szczepu należy więc zwrócić szczególną uwagę na to, aby był pozbawiony genów tych enzymów. Ponadto redukcyjne środowisko cytoplazmy uniemożliwia wprowadzenie do cząsteczki mostków dwusiarczkowych. W związku z tym białka zawierające w strukturze tego typu wiązania nie uzyskają odpowiedniej konformacji. Rozwiązaniem tego problemu może być zastosowanie szczepów zawierających mutacje w genach trxB i/lub gor, kodujących odpowiednio reduktazę tioredoksyny i glutationu. Enzymy te są odpowiedzialne za utrzymywanie tioredoksyny i glutaredoksyny w stanie zredukowanym, uniemożliwiając tworzenie mostków S-S w cytoplazmie komórek E. coli. Szczepy pozbawione wymienionych reduktaz, takie jak Origami i AD494 mają zdolność tworzenia mostków dwusiarczkowych w cytoplazmie. Największym problemem w lokalizacji rekombinowanych białek w cytoplazmie komórek bakteryjnych jest tworzenie ciał inkluzyjnych [21,28].
Periplazma
Każde rekombinowane białko, które ma zostać umiejscowione w periplazmie, musi mieć N-końcową sekwencję sygnałową, kierującą do tej przestrzeni komórkowej. Na ogół to 18-30-aminokwasowa sekwencja, składająca się z kilku aminokwasów zasadowych na końcu aminowym, hydrofobowego rdzenia i hydrofilowego końca karboksylowego rozpoznawanego przez swoiste proteazy. Enzymy te są zlokalizowane w błonie wewnętrznej, a ich centrum aktywne znajduje się po stronie periplazmatycznej. Sekwencja sygnałowa jest odcinana od łańcucha polipeptydowego podczas translokacji przez błonę wewnętrzną. Proces ten zachodzi z udziałem specyficznych transporterów błonowych (translokonu Sec lub Tat). Sekwencje sygnałowe kierujące rekombinowane białko do periplazmy pochodzą najczęściej z bakteryjnych białek OmpT, OmpA, PelB, PhoA, MalE, LamB lub β-laktamazy [5,13,16,21,26,33].
Do periplazmy często kierowane są białka mające w swojej strukturze liczne mostki dwusiarczkowe. Dzięki utleniającemu środowisku oraz obecności białek Dsb (oksydoreduktazy tiolowo-disiarczkowe) – DsbA i DsbC, możliwe jest poprawne rozmieszczenie tego typu wiązań w obrębie cząsteczki. Fuzja lub koekspresja rekombinowanego białka z Dsb może zwiększyć wydajność tworzenia wiązań – S-S – [16,21,26,33].
Białka zlokalizowane w periplazmie można łatwo uwolnić z komórek z zastosowaniem szoku osmotycznego, a otrzymany preparat jest tylko w niewielkim stopniu zanieczyszczony białkami bakteryjnymi. Ponadto ryzyko proteolitycznej degradacji polipeptydów w przestrzeni periplazmatycznej jest niewielkie, co w dużym stopniu wpływa na ilość oczyszczanych białek. Jednakże transport białek do periplazmy nie zawsze jest wydajny. Istnieje zjawisko tzw. zapychania błon, które znacznie ogranicza ten proces [25]. Można temu przeciwdziałać przez zmniejszenie poziomu produkcji heterologicznych białek. Obniża to jednak pulę syntetyzowanych polipeptydów. Innym sposobem jest koekspresja genów kodujących elementy systemu translokacji. Niektóre białka mogą tworzyć również ciała inkluzyjne w periplazmie [16].
Sekrecja na zewnątrz komórki
Pozbawione patogenności laboratoryjne szczepy E. coli mają znacznie upośledzony system sekrecji białek [15]. W związku z tym nie mają zdolności efektywnego transportowania polipeptydów przez błonę zewnętrzną. Sekrecję białek do podłoża uzyskuje się przez destabilizację struktury błony zewnętrznej wykorzystując wewnątrzkomórkowe białka lityczne. Zwiększenie przepuszczalności błony za pomocą czynników mechanicznych, enzymatycznych lub chemicznych również prowadzi do biernego transportu białek poza komórkę [21,26].
Innym sposobem jest stosowanie odpowiednio zmodyfikowanych szczepów E. coli. W tym celu stosuje się komórki pozbawione elementów strukturalnych błony zewnętrznej lub bakterie zawierające geny kodujące elementy strukturalne mechanizmów sekrecyjnych pochodzące od patogennych szczepów. Aparaty takie umożliwiają transport polipeptydu poprzez kanał białkowy bezpośrednio z cytoplazmy na zewnątrz komórki (przez błonę wewnętrzną i zewnętrzną). Kolejną strategią jest zastosowanie szczepów pozbawionych błony zewnętrznej i otoczki mureinowej. Wówczas białka mające sekwencję sygnałową kierującą do periplazmy są z powodzeniem wydzielane do podłoża hodowlanego [26].
Niektóre białka kierowane do periplazmy są w określonych warunkach wydzielane do pożywki. Jest to prawdopodobnie proces bierny, związany z jednoczesną degradacją błony zewnętrznej wywołaną wiekiem komórek lub zmianą warunków hodowli.
Sekrecja do podłoża pozwala na uzyskanie preparatów białkowych o wysokim stopniu czystości. Mimo to ten sposób produkcji rekombinowanych białek jest najrzadziej stosowany ze wszystkich dostępnych.
Podsumowanie
Na efektywność produkcji rekombinowanych białek wpływa wiele czynników – począwszy od wyboru wektora, poprzez modyfikacje genu białka docelowego, na właściwościach komórek gospodarza kończąc. W zależności od rodzaju produkowanego białka, jego właściwości, docelowej lokalizacji i zastosowania, konieczny jest dobór odpowiednich procedur. Pozwala to na uzyskanie aktywnych biologicznie białek w krótkim czasie i w satysfakcjonujących ilościach.
PIŚMIENNICTWO
[1] Abdullah N., Chase H.A.: Removal of poly-histidine fusion tags from recombinant proteins purified by expanded bed adsorption. Biotechnol. Bioeng., 2005; 92: 501-513
[PubMed]
[2] Arnau J., Lauritzen C., Petersen G.E., Pedersen J.: Current strategies for the use of affinity tags and tag removal for the purification of recombinant proteins. Protein Expr. Purif., 2006; 48: 1-13
[PubMed]
[3] Baneyx F., Recombinant protein expression in Escherichia coli. Curr. Opin. Biotechnol., 1999; 10: 411-421
[PubMed]
[4] Buchowicz J.: Biotechnologia molekularna. Modyfikacje genetyczne, postępy, problemy. Wydawnictwo Naukowe PWN, Warszawa, 2009
[5] Choi J.H., Lee S.Y.: Secretory and extracellular production of recombinant proteins using Escherichia coli. Appl. Microbiol. Biotechnol., 2004; 64: 625-635
[PubMed]
[6] Chou C.P.: Engineering cell physiology to enhance recombinant protein production in Escherichia coli. Appl. Microbiol. Biotechnol., 2007; 76: 521-532
[PubMed]
[7] Dyson M.R., Shadbolt S.P., Vincent K.J., Perera R.L., McCafferty J.: Production of soluble mammalian proteins in Escherichia coli: identification of protein features that correlate with successful expression. BMC Biotechnol., 2004; 4: 32
[PubMed] [Full Text HTML] [Full Text PDF]
[8] Gołda S., Kucharzewska P., Cisowski J., Florczyk U., Zagórska A., Jaźwa A., Łoboda A., Józkowicz A., Dulak J.: Regulacja ekspresji genów w wektorach plazmidowych: system zależny od doksycykliny i regulowany przez niedotlenienie. Biotechnol., 2007; 3: 82-97
[Full Text PDF]
[9] Gustafsson C., Govindarajan S., Minshull J.: Codon bias and heterologous protein expression. Trends Biotechnol., 2004; 22: 346-353
[PubMed]
[10] Jana S., Deb J.K.: Strategies for efficient production of heterologous proteins in Escherichia coli. Appl. Microbiol. Biotechnol., 2005; 67: 289-298
[PubMed]
[11] Jenny R.J., Mann K.G., Lundblad R.L.: A critical review of the methods for cleavage of fusion proteins with thrombin and factor Xa. Protein Expr. Purif., 2003; 31: 1-11
[PubMed]
[12] Kamionka M.: Engineering of therapeutic proteins production in Escherichia coli. Curr. Pharma. Biotechnol., 2011; 12: 268-274
[PubMed] [Full Text HTML] [Full Text PDF]
[13] Lee S.F., Li Y.J., Halperin S.A.: Overcoming codon-usage bias in heterologous protein expression in Streptococcus gordonii. Microbiol., 2009; 155: 3581-3588
[PubMed] [Full Text HTML] [Full Text PDF]
[14] Li W., Zhou X., Lu P.: Bottlenecks in the expression and secretion of heterologous proteins in Bacillus subtilis. Res. Microbiol., 2004; 155: 605-610
[PubMed]
[15] Mergulhao F.J., Summers D.K., Monteiro G.A.: Recombinant protein secretion in Escherichia coli. Biotechnol. Adv., 2005; 23: 177-202
[PubMed]
[16] Ming Y.M., Wei Z.W., Lin C.Y., Sheng G.Y.: Development of a Bacillus subtilis expression system using the improved Pglv promotor. Microb Cell Fact., 2010; 9: 55
[PubMed] [Full Text HTML] [Full Text PDF]
[17] Ni Y., Chen R.: Extracellular recombinant protein production from Escherichia coli. Biotechnol. Lett., 2009; 31: 1661-1670
[PubMed]
[18] Nuc P., Nuc K.: Produkcja rekombinowanych białek w Escherichia coli. Postępy Biochem., 2006; 52: 448-456
[PubMed]
[19] Prior J.E., Lynch M.D., Gill R.T.: Broad-host-range vectors for protein expression across Gram negative hosts. Biotechnol. Bioeng., 2010; 106: 326-332
[PubMed]
[20] Rai M., Padh H.: Expression systems for production of heterologous proteins. Curr. Sci., 2001; 80: 1121-1128
[21] Redwan E.L.: The optimal gene sequence for optimal protein expression in Escherichia coli: principle requirements. Arab. J. Biotechnol., 2006: 9: 493-510
[22] Sahdev S., Khattar S.K., Saini K.S.: Production of active eukaryotic proteins through bacterial expression systems: a review of the existing biotechnology strategies. Mol. Cell. Biochem., 2008: 307: 249-264
[PubMed]
[23] Sęktas M.: Ekspresja genów klonowanych w wektorach plazmidowych w zrekombinowanych szczepach Echcerichia coli. Kosmos, 2002; 51: 365-373
[Full Text PDF]
[24] Sorensen H.P.: Towards universal systems for recombinant gene expression. Microb. Cell. Fact., 2010; 9: 27
[PubMed] [Full Text HTML] [Full Text PDF]
[25] Sorensen H.P., Mortensen K.K.: Advanced genetic strategies for recombinant protein expression in Escherichia coli. J. Biotechnol., 2005; 115: 113-128
[PubMed]
[26] Sorensen H.P., Mortensen K.K.: Soluble expression of recombinant proteins in the cytoplasm of Escherichia coli. Microb. Cell. Fact., 2005; 4: 1
[PubMed] [Full Text HTML] [Full Text PDF]
[27] Staroń A., Grabowska A., Jagusztyn-Krynicka E.K.: Nadprodukcja i oczyszczanie rekombinowanych, heterologicznych białek w komórkach Escherichia coli. Postępy Mikrobiol., 2008; 47: 83-95
[Full Text PDF]
[28] Tate C.G., Haase J., Baker C., Boorsma M., Magnani F., Vallis Y., Williams D.C.: Comparison of seven different heterologous protein expression systems for the production of the serotonin transporter. Biochim. Biophys. Acta, 2003; 1610: 141-153
[PubMed]
[29] Terpe K.: Overview of bacterial expression systems for heterologous protein production: from molecular and biochemical fundamentals to commercial systems. Appl. Microbiol. Biotechnol., 2006; 72: 211-222
[PubMed]
[30] Terpe K.: Overview of tag protein fusions: from molecular and biochemical fundamentals to commercial systems. Appl. Microbiol. Biotechnol., 2003; 60: 523-533
[PubMed]
[31] Waugh D.S.: Making the most of affinity tags. Trends. Biotechnol., 2005; 23: 316-320
[PubMed]
[32] Wong L.S.: Advances in the use of Bacillus subtilis for the expression and secretion of heterologous proteins. Curr. Opin. Biotechnol., 1995; 6: 517-522
[PubMed]
[33] Yoon S.H., Kim S.K., Kim J.F.: Secretory production of recombinant proteins in Escherichia coli. Recent. Pat. Biotechnol., 2010; 4: 23-29
[PubMed]
Autorzy deklarują brak potencjalnych konfliktów interesu.
